RNA二级结构中有AU、GC、GU三种碱基配对模式。连续的碱基配对相互堆积而构成茎(stem),茎在三维空间中呈现出一段双螺旋结构,中间若出现少数不配对的碱基则会形成凸环(bulge loop)或内环(interior loop)。相邻连续的一段序列因两端互补而回折,就会形成像发卡一样的结构,连茎带环形象地称之为发卡环(hairpin loop)。连接各发卡环而未能配对的区域则叫做多分支环(multi-branched loop),序列末端没有形成配对的单链叫做自由单链(unstructured single strand)。这些基本结构单元又可称之为RNA二级结构的基序(motif)。
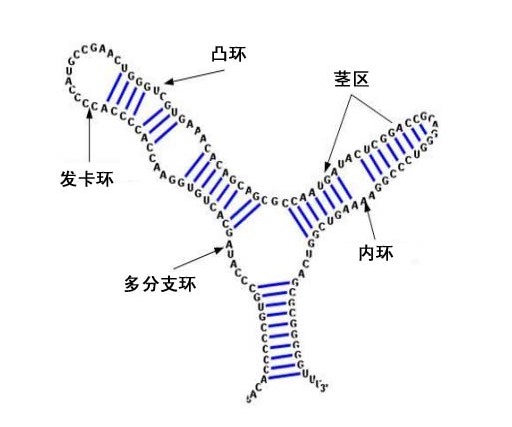 图1. RNA二级局部结构茎区及环的示意图。
图1. RNA二级局部结构茎区及环的示意图。
MicroRNA局部结构的识别方法大致可以分为3类:基于序列或结构保守性的方法、基于实验数据驱动以及基于机器学习的方法。
在RNA分子中,结构保守性一般大于序列的保守性。如绝大多数tRNA分子的二级结构都是三叶草型结构,三级结构呈倒L型,而它们的核苷酸序列却存在部分差异。比较序列分析法可以按照序列比对与结构预测的先后顺序分为三种:先比对后预测,如Pfold;先预测后比对,如MARNA;结构预测与序列比对同时进行,如RNAStructure。
基于新实验数据方法的提出得益于“深度测序”技术的发展,通过生物信息学工具对大规模的序列数据进行处理、注释以及新的特征描述,从而发现新的MicroRNA序列。
MicroRNA的序列和结构特征的机器学习方法,基于阳性和阴性的MicroRNA样本构建分类器预测未知序列。如输入待测MicroRNA的一组特征(发夹等),输出1或0表示候选对象是否是MicroRNA。机器学习的算法一般包括支持向量机(SVM)、神经网络、隐型马尔可夫模型(HMM)和朴素贝叶斯模型(NB)等。
安必奇生物提供以上三种方法对MicroRNA局部可杂交的区域,如联合序列、内环、10个或10个以上连续核苷酸的发夹结构等进行合理的预测分析。已有研究表明,高度保守的基序是MircroRNA Agomir/ Antagomir的良好靶标,而可变的局部基序可能会导致非序列特异性效应,所以预测RNA二级局部结构中的高度保守的基序,并把它们作为靶标,是提高MircroRNA Agomir/ Antagomir命中率的有力方式。
如果您想知道更多相关的服务信息,请通过热线电话或是E-mail ()与我们联系,我们的技术人员将在24小时内回复您的所有问题及要求。
24小时服务在线

